DNA-Replikation: Hinweise zu DNA-Replikation, Reparatur und Rekombination
Hinweise zur DNA-Replikation, Reparatur und Rekombination!
DNA Replikation:
Halbkonservative DNA-Replikation:
DNA-Replikation ist eine autokatalytische Funktion von DNA. Es tritt normalerweise während der S-Phase des Zellzyklus auf, wenn Chromosomen in stark ausgedehnter Form vorliegen. Wie von Watson und Crick vorgeschlagen, ist die DNA-Replikation halbkonservativ.
Bei der halbkonservativen Replikation würden sich die beiden Stränge voneinander trennen, ihre Integrität beibehalten und jeder wird aus dem Pool der Nukleotide seinen komplementären Strang synthetisieren. Das Ergebnis wäre, dass das neu synthetisierte Molekül einen der beiden Stränge des Stammmoleküls tragen oder konservieren würde und der andere Strang neu zusammengefügt würde. Es gibt hinreichende Beweise, um zu beweisen, dass sich doppelsträngige DNA tatsächlich auf halbkonservative Weise repliziert.

Meselson und Stahls Experiment (1958):
Diese Arbeiter kultivierten Escherichia coli in einem Medium, das 15 N Isotope enthielt. Nachdem diese Generationen in diesem Medium repliziert worden waren, enthielten beide Stränge dieser DNA 15 N als Bestandteile von Purinen und Pyrimidinen. Wenn diese Bakterien mit 15 N in ein Kulturmedium überführt wurden, das 14 N enthielt, wurde festgestellt, dass DNA, die von der frischen Bakteriengeneration abgetrennt wurde, einen Strang besitzt, der schwerer ist als der andere.
Der schwerere Strang stellt den elterlichen Strang dar und der leichtere ist der neue, der aus dem Kulturmedium synthetisiert wird. Dies weist auf eine halbkonservative Methode der DNA-Replikation hin und schließt sowohl konservative als auch dispersive Modelle der DNA-Synthese und -Replikation aus.
Konservative Replikation würde keine DNA-Moleküle mit "Hybrid" -Konstitution erzeugen. Wenn die Replikation dispersiv wäre, hätte sich die DNA durch jede Generation von „schwer“ zu „leicht“ verschoben.
Nachfolgende Studien haben die Schlussfolgerung von Meselson und Stahl bestätigt, dass die DNA-Replikation halbkonservativ ist, und sie auf viele andere Organismen, einschließlich höherer Pflanzen und Tiere, ausgeweitet.
Cairns Autoradiographie-Experiment:
Er verwendete radioaktives Thymin oder tritiiertes Thymidin. Durch das Züchten von E. coli in einem Kulturmedium, das tritiiertes Thymidin enthielt, wurde die Radioaktivität in die Tochter-DNA-Moleküle eingebaut.
In der Autoradiographie zeigt das duplizierte DNA-Molekül eine replizierende Gabel, den Punkt, an dem zwei Ketten zu vier werden. Nach der ersten Replikation wird gesehen, dass die Radioaktivität nur in einen der DNA-Stränge eingebaut ist, und es wird gefunden, dass beide Stränge nach der zweiten Replikation markiert werden. Dies unterstützt die halbkonservativen Modi der DNA-Replikation.
JH Taylors Experimente an Viciafaba-Wurzelspitzen (1957) bestätigen auch die halbkonservative Methode der DNA-Replikation.
ich. Diskontinuierliche DNA-Replikation:
Okazaki schlug vor, dass die DNA-Synthese gleichzeitig auf beiden DNA-Strängen abläuft, wobei das gleiche Enzym DNA-Polymerase in Form kleiner isolierter Fragmente verwendet wird. Diese Segmente sind als Okazaki-Stücke bekannt und bestehen aus 1000-2000 Nukleotiden. Diese werden durch das Enzym Polynukleotidligase miteinander verbunden, wodurch die Bildung der Polynukleotidkette abgeschlossen wird.
Die diskontinuierliche Synthese von DNA wird durch ein Autoradiogramm unterstützt.

ii. Unidirektionale und bidirektionale Replikation von DNA:
J. Cairns aus seinen Experimenten schlussfolgerte, dass die DNA-Synthese an einem festen Punkt auf den Chromosomen beginnt und in eine Richtung verläuft, aber neuere Experimente legen die bidirektionale Replikation nahe.
Levinthal und Cairns schlugen vor, dass sich die beiden Stränge während der Replikation vor der Replikation nicht vollständig trennen. Stattdessen beginnen sie an einem Ende zu entpacken und gleichzeitig ziehen die entpackten Segmente ihre Nukleotidpaare an. Auf diese Weise gehen das Entpacken der ursprünglichen DNA-Stränge und die Synthese frischer DNA-Stränge nebeneinander.

DNA-Polymerase:
DNA-Polymerase ist das Hauptenzym der DNA-Replikation. Seine Aktivität wurde erstmals von Kornberg im Jahr 1956 demonstriert. Sie katalysiert die kovalente Addition von Desoxyribonukleotiden an das 3'-OH eines bereits existierenden Nukleotids, das als Primer bezeichnet wird.
DNA-Polymerase-1-Enzym wird jetzt als DNA-Reparaturenzym und nicht als Replikationsenzym betrachtet. Es ist bekannt, dass dieses Enzym fünf aktive Zentren aufweist, nämlich die Template-Stelle, die Primer-Stelle, die 5 '-> 3'-Schnittstelle oder die Exonuklease-Stelle, die Nucleosidtriphosphat-Stelle und die 3' -> 5'-Schnittstelle (oder die 3 '-> 5'-Exonuklease-Stelle ).
DNA-Polymerase I:
Es ist hauptsächlich daran beteiligt, RNA-Primer aus Okazaki oder Vorläuferfragmenten zu entfernen und die entstehenden Lücken aufgrund seiner 5 '-> 3'-Polymerisationskapazität zu füllen. Das DNA-Polymerase-I-Enzym kann auch Thymin-Dimere entfernen, die durch UV-Bestrahlung entstehen, und die Lücke durch Herausschneiden füllen. Dies wird als Korrekturlesung oder Editierfunktion dieses Enzyms bezeichnet.
DNA-Polymerase-II:
Dieses Enzym ähnelt der DNA-Polymerase-I in seiner Aktivität, ist jedoch ein DNA-Reparaturenzym und bewirkt das Wachstum in 5 '-> 3'-Richtung unter Verwendung freier 3'-OH-Gruppen.
DNA-Polymerase-III:
Es spielt eine wesentliche Rolle bei der DNA-Replikation. Es ist ein multimeres Enzym oder Holoenzym mit zehn Untereinheiten wie α, β, ε, θ, г, y, δ, δ ', x und. Alle diese zehn Untereinheiten sind für die DNA-Replikation in vitro erforderlich. Alle haben jedoch unterschiedliche Funktionen. Beispielsweise hat eine α-Untereinheit 3 '-> 5'-Exonuklease-Korrekturlesungs- oder Editieraktivitäten. Das Kernenzym umfasst drei Untereinheiten - α, β und θ. Die verbleibenden sieben Untereinheiten erhöhen die Prozessivität (Prozessivität bedeutet Schnelligkeit und Effizienz, mit der eine DNA-Polymerase die Wachstumskette verlängert).
Eukaryotische DNA-Polymerase:
Es wurde gefunden, dass Eukai-Yotes (z. B. Hefe, Rattenleber, menschliche Tumorzellen) die folgenden fünf Arten von DNA-Polymerasen enthalten:
(i) DNA-Polymerase α:
Dieses Enzym mit relativ hohem Molekulargewicht wird auch als cytoplasmatische Polymerase oder große Polymerase bezeichnet. Es kommt sowohl im Zellkern als auch im Zytoplasma vor.
(ii) DNA-Polymerase β:
Dieses Enzym wird auch als Kernpolymerase oder kleine Polymerase bezeichnet und kommt nur in Wirbeltieren vor.
(iii) DNA-Polymerase y:
Dieses Enzym wird als mitochondriale Polymerase bezeichnet und ist im Kern kodiert.
(iv) DNA-Polymerase δ:
Dieses Enzym wird in Säugetierzellen gefunden und ist für die DNA-Synthese-Prozessivität PCNA-abhängig (PCNA = Proliferationszellkern-Antigen).
(v) DNA-Polymerase & epsi;
Es war zuvor als DNA-Polymerase 5II bekannt. Dieses Enzym ist PCNA-unabhängig und kommt in HeLa-Zellen von Säugetieren und aufstrebenden Hefen vor.
Bei der großen DNA-Polymerase a handelt es sich bei dem vorherrschenden DNA-Polymerase-Enzym um eukaryontische Zellen, und es wurde angenommen, dass es lange Zeit das einzige Enzym war, das an der DNA-Replikation beteiligt ist. Nun ist jedoch eine weitere Polymerase, nämlich die DNA-Polymerase 8, an der eukaryotischen DNA-Replikation beteiligt.
DNA-Primer:
Diese Enzyme katalysieren die Synthese von RNA-Primern, die eine Voraussetzung für die Initiierung der DNA-Replikation in der überwiegenden Mehrheit der Organismen sind. Bevor die eigentliche DNA-Replikation beginnt, müssen kurze RNA-Oligonukleotidsegmente, kurz RNA-Primer oder einfach als Primer bezeichnet, mit einem DNA-Primase-Enzym unter Verwendung von Ribonukleosidtriphosphaten synthetisiert werden.
Dieser RNA-Primer wird durch Kopieren einer bestimmten Basensequenz aus einem DNA-Strang synthetisiert und unterscheidet sich von einem typischen RNA-Molekül darin, dass der Primer nach der Synthese an die DNA-Matrize mit Wasserstoff gebunden bleibt.
Die Primer sind in Eukaryonten etwa 10 Nukleotide lang und werden in Intervallen auf dem nacheilenden Strang hergestellt, wo sie durch das DNA-Polymerase-Enzym verlängert werden, um jedes Okazaki-Fragment zu beginnen. Diese RNA-Primer werden später mit Hilfe des DNA-Reparatursystems in Eukaryoten ausgeschnitten und mit DNA gefüllt.
In Bakterien sind zwei verschiedene Enzyme dafür bekannt, Primer-RNA-Oligonukleotide zu synthetisieren - RNA-Polymerase (am führenden Strang) und DNA-Primase (am verzögernden Strang).
Polynukleotid-Ligase:
Dieses Enzym ist sowohl bei der DNA-Replikation als auch bei der DNA-Reparatur ein wichtiges Enzym. DNA-Ligase katalysiert die Bildung einer Phosphodiester-Bindung zwischen der 5'-Phosphorylgruppe eines Nukleotids und der 3-OH-Gruppe des unmittelbaren Nachbarn an der Seite eines Nick in einem DNA-Strang; so versiegelt es die Kerben in einem DNA-Strang.
Endonukleasen:
Endonucleasen, insbesondere Restriktionsendonucleasen, sind auch während der DNA-Replikation sowie der DNA-Reparatur wichtig. Während der DNA-Replikation kann eine Endounclease einen Ursprung im Ursprung erzeugen, um die Replikation zu initiieren, oder sie kann Kerben induzieren, um einen Drehpunkt zu erzeugen, der das Abwickeln der DNA erleichtert.
Enzyme, die an der Öffnung der DNA-Helix beteiligt sind:
DNA-Helikasen:
DNA-Helikasen sind ATP-abhängige Abwicklungsenzyme, die die Trennung der beiden Elternstränge fördern und Replikationsgabeln einrichten, die sich schrittweise vom Ursprung entfernen. Das Abwickeln der Templat-DNA-Helix an einer Replikationsgabel könnte prinzipiell durch zwei aufeinander abgestimmte DNA-Helikasen katalysiert werden, von denen eine entlang des Führungsstrangs und die andere entlang des nacheilenden Strangs verläuft.
Helix-destabilisierender Strang (auch Einzelstrang-DNA-Bindungsproteine oder SSBPs genannt):
Hinter der Replikationsgabel werden die einzelnen DNA-Stränge durch die Wirkung von SSB-Proteinen daran gehindert, umeinander herumzuwickeln (oder doppelsträngige Haarnadelschleifen in jedem einzelnen Strang zu bilden). SSB-Proteine binden an freiliegende DNA-Stränge, ohne die Basen zu überdecken, die daher für den Templating-Prozess verfügbar bleiben.
Topoisomerasen (DNA-Gyrasen):
Die Wirkung einer Helikase führt vor der Replikationsgabel eine positive Superspule in die Duplex-DNA ein. Enzyme, so genannte Topoisomerasen, entspannen die Superspule, indem sie sich an den transienten Superspiralen-Duplex anheften, einen der Stränge einschneiden und ihn durch den ungebrochenen Strang drehen. Der Nick wird dann wieder versiegelt.
Ein Typ von Topoisomerase (dh Topoisomerase I) verursacht einen einsträngigen Bruch oder Einschnitt, der es den beiden Abschnitten der DNA-Helix auf beiden Seiten des Einschnittes ermöglicht, sich frei zueinander zu drehen, wobei die Phosphodiesterbindung in dem dem Einschnitt gegenüberliegenden Strang als verwendet wird ein Drehpunkt. Ein zweiter Typ von Topoisomerase (dh Topoisomerase II) bildet gleichzeitig eine kovalente Bindung an beide DNA-Helix-Stränge, wodurch ein vorübergehender Doppelstrangbruch in der Helix entsteht.
Replikon:
Ein Replikon ist die Einheit der DNA, in der einzelne Replikationsakte stattfinden, das heißt, es ist zur DNA-Replikation unabhängig von anderen DNA-Abschnitten fähig. Daher hat jedes Replikon einen Ursprung, von dem aus die Replikation initiiert wird, und es kann einen Terminus haben, an dem die Replikation beendet wird.
Die bakteriellen und viralen Chromosomen enthalten normalerweise ein einzelnes Replikon / Chromosom. Der Phage T hat zwar zwei Ursprünge, einen primären und einen sekundären, jedoch ist der sekundäre Ursprung bei Vorliegen eines Primärursprungs in der Regel nicht funktionell. In E. coli wird der Ursprungsort als genetischer Locus oriC identifiziert.
Ein vollständiger prokaryotischer Ursprung unterstützt die folgenden drei Funktionen: (1) Initiierung der Replikation, (2) Kontrolle der Häufigkeit von Initiationsereignissen und (3) Segregation der replizierten Chromosomen in die Tochterzellen.
Ursprünge wurden in Bakterien, Hefen, Chloroplasten und Mitochondrien festgestellt; Ihr wesentliches allgemeines Merkmal ist, dass sie A: T-reich sind, was für ein leichteres Abwickeln während der Replikation wichtig sein kann. Der bakterielle Ursprung enthält viele verschiedene kurze (<10 bp) Stellen, die für seine Funktion benötigt werden. Diese Standorte sind manchmal durch bestimmte Abstände voneinander getrennt, jedoch nicht durch spezifische Sequenzen. Diese spezifisch lokalisierten Stellen werden für die Bindung verschiedener Proteine benötigt, die an der DNA-Replikation beteiligt sind.
Mehrere prokaryotische Replikons haben spezifische Stellen, die als Terminus bezeichnet werden, die die Replikationsgabelbewegung stoppen und dadurch die DNA-Replikation beenden. Das E. coli-Chromosom hat zwei Termini, die als T 1 und T 2 bezeichnet werden und sich auf beiden Seiten des Punkts befinden, an dem sich die Replikationsgabeln treffen würden. Jede Endstation ist spezifisch für eine Richtung der Gabelbewegung.
Die T 1 und T 2 sind so angeordnet, dass jede Gabel die andere passieren muss, um den für sie spezifischen Endpunkt zu erreichen. Die Replikationstermination erfordert das Produkt des tus-Gens, das wahrscheinlich für ein Protein kodiert, das T und T 2 erkennt.
In Eukaryoten hat jedes Chromosom mehrere Replikons (z. B. 500 Hefe; Drosophila 3500; Maus 25.000; Viciafaba 35.000). Zu irgendeinem Zeitpunkt während der S-Phase durchlaufen nur einige dieser Replikons eine Replikation. Jedes Replikon scheint zu einem bestimmten Zeitpunkt in einer bestimmten Reihenfolge aktiviert zu sein. Die Länge eines eukaryotischen Replikons kann von 40 Kb in Hefe und Drosophila bis etwa 300 Kb in Vicia variieren.
Die Anzahl der nachweisbaren Replikons scheint mit dem Entwicklungsstadium und dem Zell- oder Gewebetyp zu variieren. Dies wird auf der Grundlage gewebespezifischer Ursprünge erklärt, so dass einige Ursprünge in einigen Geweben aktiv sind, während andere in einigen anderen Geweben aktiv sind.
Dies wird am Beispiel von Drosophila veranschaulicht, bei dem die frühen embryonalen Zellen zehnmal so viele Replikons aufweisen wie die adulten somatischen Zellen. Die verfügbaren Beweise legen nahe, dass eukaryotische Replikons keine Termini haben.
Treue der Replikation:
Die Fehlerrate der DNA-Replikation ist viel niedriger als die der Transkription, da die Bedeutung der genetischen Botschaft von Generation zu Generation erhalten werden muss. Beispielsweise beträgt die spontane Mutationsrate in E. coli etwa einen Fehler pro 10 10 Basen, die während der Replikation aufgenommen wurden.
Dies ist hauptsächlich auf das Vorhandensein der kleineren tautomeren Formen der Basen zurückzuführen, die veränderte Basenpaarungseigenschaften aufweisen. Die Fehlerrate wird durch verschiedene Mechanismen minimiert. DNA-Polymerasen bauen ein eingehendes Nucleotid nur dann ein, wenn es mit dem Templat-Nucleotid in seinem aktiven Zentrum das richtige Basenpaar bildet.
Der gelegentliche Fehler wird durch die mit der Polymerase assoziierte 3 '-> 5'-Korrektur-Exonuklease festgestellt. Dadurch wird das falsche Nucleotid vor einer weiteren Inkorporation vom 3'-Ende entfernt, wodurch die Polymerase die korrekte Base einführen kann. Damit das Korrekturlesen von Exonuclease ordnungsgemäß funktioniert, muss es in der Lage sein, ein korrektes Basenpaar von einem falschen zu unterscheiden.
Die erhöhte Mobilität von "nicht verankerten" Basenpaaren am 5'-Ende von neu initiierten DNA-Fragmenten mit verzögertem Strang führt dazu, dass sie niemals korrekt erscheinen und daher nicht korrigiert werden können. Daher sind die ersten wenigen Nukleotide Ribonukleotide (RNA), so dass sie anschließend als Material mit geringer Genauigkeit identifiziert werden können und durch DNA ersetzt werden können, die aus dem benachbarten Fragment länglich (und durch Korrektur gelesen) wird. Fehler, die dem Korrekturlesen entgehen, werden durch einen Mismatch-Reparaturmechanismus korrigiert.
Mechanismus der DNA-Replikation in Prokaryoten:
In-vitro-DNA-Replikation wurde ausführlich in E. coli sowie in den Phagen und Plasmiden von E. coli untersucht. In E. coli umfasst der Prozess der DNA-Replikation die folgenden drei Hauptschritte:
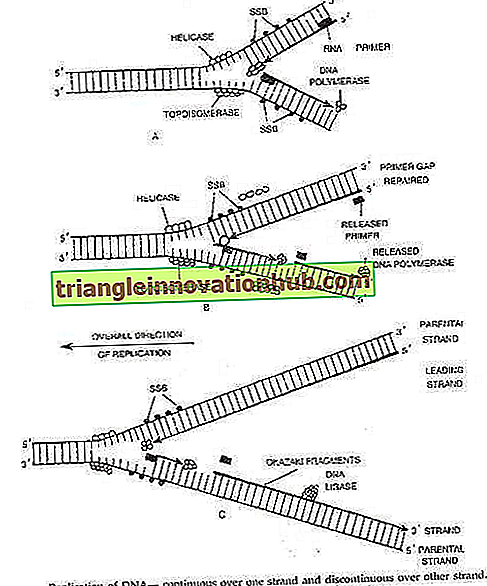
1. Initiierung der DNA-Replikation:
Dieses Verfahren umfasst drei Schritte: (i) Erkennen des Ursprungs (O), (ii) Öffnen von DNA-Duplex zur Erzeugung einer Region von einzelsträngiger DNA und (iii) Einfangen von Dna-B-Protein (dh 5'3'-Helikase) fungiert auch als Aktivator für Primase). Somit bindet der Dna-A- (oder Initiatorprotein) -ATP-Komplex bei 9 bp invertierten Wiederholungsregionen (R,, R,, R v R 4 ) von ori C von E. coli und fördert die Öffnung des DNA-Duplex in einer Region von drei direkte Wiederholungen der 13-bp-Sequenz (13-mers genannt).
Die Öffnung erfolgt von rechts 13 m nach links und erfordert negativ geschliffene DNA und HU- oder IHF-Initiatorproteine. Dna B (Helicase) wird auf freiliegende einzelsträngige DNA übertragen und bewirkt ein Abwickeln der DNA in Gegenwart von A TP, SSB-Protein und DNA-Gyrase (eine Topoisomerase).
Dies führt zum Abwickeln des DNA-Duplex und die Replikation von ori C erfolgt in beide Richtungen (bidirektional). Die SSB-Bindung tritt an einzelsträngigen Regionen auf und zwei Dna B-Komplexe (= Primosomen) werden auf jeden Strang geladen.
2. Dehnung der DNA-Kette:
Dieser Schritt erfordert die Anwesenheit der folgenden Enzyme und Faktoren: 1. Dna B oder Helicase (auch mobiler Promotor genannt); 2. Primase (Dna G); 3. DNA-Polymerase-Holoenzym (oder DNA pol III HE); 4. SSB-Protein; 5. RNAase H, die RNA-Primer entfernt; 6. DNA-Polymerase I, die zum Schließen der durch RNA-Primer entstandenen Lücke verwendet wird, und 7. DNA-Ligase (die primerlose Okazaki-Fragmente in einen kontinuierlichen Strang umwandelt). Während des Übergangs vom Beginn zum Dehnungsverlauf treten folgende Ereignisse auf:
(i) Wenn sich Helikase (oder Dna B) in Richtung 5 '-> 3' bewegt, erzeugt sie eine Replikationsgabel durch Öffnen des DNA-Duplex.
(ii) Der Helicase enthaltende DNA-Strang wird zum nacheilenden Strang. Die DNA-Primase verbindet sich mit der Dna-B-Helikase und bildet das Primosom, das mehrere Primer für den verzögernden Strang und den Einzel-RNA-Primer für den führenden Strang synthetisiert.
(iii) Für die Synthese eines nacheilenden Strangs muss der DNA pol III HE an dem gleichen Strang arbeiten, an den die Dna B-Helicase gebunden ist, aber sie wandert in entgegengesetzter Richtung.
(iii) DnaB-Helikase, Dna-Gprimase und DNA-pol-III-FIE arbeiten bei der Strangverlängerung zusammen. Helicase- und DNA-Polymerase-Assemblierung bleibt prozessiv, dh sie bleiben fest an die Gabel gebunden und bleiben während der gesamten Reaktion gebunden.
Die Synthese (- Dehnung) der nachlaufenden und führenden Stränge erfolgt nach etwas unterschiedlichen Methoden; Es ist weitaus komplexer als der Leitstrang:
(A) Diskontinuierliche Synthese am verzögerten Strang:
1. Primase wird aus der Lösung aufgenommen und durch Helicase (DnaB) aktiviert, um einen aRNA-Primer (10 bis 20 nt oder Nukleotide lang) auf dem nacheilenden Strang zu synthetisieren.
2. Die RNA-Primer werden von DNA pol III HE am nacheilenden Strang erkannt und zur Synthese von Vorläufer- oder Okazaki-Fragmenten verwendet. Tatsächlich wird jeder neue RNA-Primer von der gamma (y) -Untereinheit von DNA pol III HE erkannt und mit einer p-Untereinheit der gleichen Polymerase beladen. Diese vorgeladene p-Untereinheit kann dann den Kern von DNA Poly III HE einfangen, wenn sie verfügbar ist, nachdem sie ihren Syntheseauftrag für das vorangehende Okazaki-Fragment abgeschlossen hat.
3. Nach Fertigstellung der Okazaki-Fragmente werden die RNA-Primer durch DNA-Polymerase 1 ausgeschnitten, die dann die entstandenen Lücken mit DNA füllt.
4. Nachdem die DNA-Polymerase I die letzten Desoxyribonukleotide in die durch den ausgeschnittenen Primer hinterlassene Lücke hinzugefügt hat, bildet das Enzym DNA-Ligase die Phosphodiester-Bindung, die das freie 3'-Ende des Primer-Austausches mit dem 5'-Ende des Okazaki-Fragments verbindet.
(B) Kontinuierliche Synthese am Leitstrang:
1. Bei der bidirektionalen DNA-Replikation wird der führende Strang einmal auf jedem der Elternstränge vorbereitet.
2. Der RNA-Primer des führenden Strangs wird durch RNA-Polymeraseenzym synthetisiert.
3. DNA pol III HE bewirkt eine Verlängerung des Führungsstrangs und schließlich geben DNA pol 1 und Ligase-Enzyme dem Führungsstrang den letzten Schliff, wie im Fall des nacheilenden Strangs.
DNA-Replikation in Eukaryoten :
Die eukaryotische DNA-Replikation erfordert zwei verschiedene DNA-Polymerase-Enzyme, nämlich DNA-Polymerase a und DNA-Polymerase δ. DNA-Polymerase δ synthetisiert die DNA auf dem führenden Strang (kontinuierliche DNA-Synthese), während DNA-Polymerase a die DNA auf dem nacheilenden Strang synthetisiert (diskontinuierliche DNA-Synthese). Neben diesen beiden Enzymen DNA-Replikation: (1) T-Antigen; (2) Replikationsfaktor A oder RF-A (auch als RP-A oder eukaryontisches SSB bezeichnet); (3) Topoisomerase I; (4) Topoisomerase II; (5) proliferierendes zelluläres Antigen (PCNA, auch als Cyclin bezeichnet) und (6) Replikationsfaktor Cor RF-C.
Der Prozess der eukaryotischen DNA-Replikation umfasst die folgenden Schritte:
1. Vor Beginn der DNA-Synthese gibt es eine vorsynthetische Stufe von 8 bis 10 Minuten zur Bildung eines abgewickelten DNA-Komplexes. Dieser Schritt benötigt nur drei gereinigte Proteine, nämlich T-Antigen (T-ag oder Tumorantigen), RF-A und Topiosomerasen I und II.
2. Das T-Antigen bildet unter Verwendung seiner DNA-Bindungsdomäne einen Komplex mit mehreren Untereinheiten mit den Stellen I und II in Gegenwart von A TP und verursacht lokales Abwickeln.
3. Eine umfangreichere Duplex-Abwicklung tritt aufgrund der Assoziation von RF-A und einer Topoisomerase mit der Hilfe der DNA-Helikasenkomponente von T-her auf. Topoisomerasen helfen beim Abwickeln von DNA, indem sie die Topologie der DNA an der Replikationsgabel verändern.
4. RF-A- oder SSB-Proteine binden an abgewickelte einzelsträngige DNA.
5. Die Primer-RNA-Synthese wird durch Primase durchgeführt, die eng mit DNA-Polymerase ex verbunden ist.
6. DNA-Polymerase a hilft bei der Synthese eines Okazaki-Fragments in 5'- bis 3'-Richtung.
7. Replikationsfaktor C (oder RF-C) und PCNA (Cyclin) helfen beim Umschalten von DNA-Polymerasen, sodass pol a durch pol 5 ersetzt wird, das dann kontinuierlich DNA auf dem führenden Strang synthetisiert.
8. Ein anderes Okazaki-Fragment wird dann aus dem Replikationsgabel auf dem nacheilenden Strang durch einen pol a-Primase-Komplex synthetisiert, und dieser Schritt wird immer wieder wiederholt, bis das gesamte DNA-Molekül bedeckt ist.
9. Die RNA-Primer werden entfernt und die Lücken werden wie bei der prokaryotischen DNA-Replikation gefüllt.
Vor kurzem wurde die Rolle der DNA-Polymerase e bei der DNA-Replikation hervorgehoben, so dass jetzt bekannt ist, dass drei DNA-Polymerasen (a, δ und ε) an der eukaryotischen DNA-Replikation beteiligt sind. A. Sugino und Kollegen haben vorgeschlagen, dass die DNA-Polymerase a sowohl am führenden als auch am nacheilenden Strang funktionieren könnte (da die Polymerase a eine α-Primase-Aktivität aufweist), während die Polymerase e und die Polymerase 5 an der Verlängerung des vorauslaufenden bzw. des nacheilenden Strangs beteiligt sind.
DNA-Schadens- und Reparaturmechanismus
Arten von DNA-Schäden:
1. DNA-Läsionen:
Eine Veränderung der normalen chemischen oder physikalischen Struktur der DNA wird als DNA-Läsionen bezeichnet. Viele exogene Agenzien wie Chemikalien und Strahlung können Änderungen an den Positionen der Stickstoff- und Kohlenstoffatome in den heterocyclischen Ringsystemen der Basen und einigen der exocyclischen funktionellen Gruppen (dh den Keto- und Aminogruppen der Basen) verursachen.
Dies kann zu einem Verlust der Basenpaarung oder einer veränderten Basenpaarung führen (z. B. kann ein geändertes A ein Basenpaar mit C anstelle von T sein). Wenn eine solche Läsion in der DNA verbleibt, kann eine Mutation in der DNA durch direkte oder indirekte Mutagenese fixiert werden.
Alternativ kann die chemische Veränderung eine physikalische Verzerrung in der DNA erzeugen, die die Replikation und / oder Transkription blockiert und den Zelltod verursacht. Somit können DNA-Läsionen mutagen und / oder tödlich sein. Einige Läsionen sind spontan und treten aufgrund der inhärenten chemischen Reaktivität der DNA und der Anwesenheit normaler, reaktiver chemischer Spezies in der Zelle auf.
Zum Beispiel wird das Cytosin der Base spontan hydrolysiert, um Uracil zu erhalten. Wenn es nicht repariert wird, bildet das resultierende Uracil während der nachfolgenden Replikation ein Basenpaar mit Adenin, was zu einer Punktmutation führt. Die Depurinierung ist eine weitere spontane Hydrolysereaktion, die die Spaltung der N-glycosylischen Bindung zwischen N-9 der Purinbasen A und G und C-1 'des Desoxyribosezuckers und somit den Verlust von Purinbasen aus der DNA beinhaltet. Das Zucker-Phosphat-Rückgrat der DNA bleibt intakt. Die resultierende apurinische Stelle ist eine nicht kodierende Läsion, da in den Purinbasen kodierte Information verloren geht.
2. Oxidationsschaden:
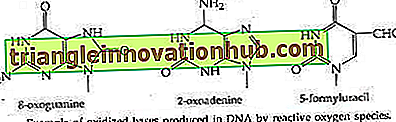
Dies geschieht unter normalen Bedingungen aufgrund der Anwesenheit reaktiver Sauerstoffspezies (ROS) in allen aeroben Zellen, beispielsweise Superoxid, Wasserstoffperoxid und vor allem dem Hydroxylradikal (OH). Dieses Radikal kann die DNA I an mehreren Stellen angreifen und eine Reihe von Oxidationsprodukten mit veränderten II-Eigenschaften erzeugen, beispielsweise 8-Oxoguanin, 2-Oxoadenin und 5-Formyluracil. Deren Mengen können durch Hydroxylradikale aus der durch ionisierende Strahlung verursachten Radiolyse von Wasser erhöht werden.
3. Alkylierung:
Alkylierungsmittel sind elektrophile Chemikalien, die leicht Alkylgruppen (z. B. Methylgruppen) an verschiedene Positionen an Nukleinsäuren anfügen, die sich von denen unterscheiden, die durch normale Methylierungsenzyme methyliert werden. Übliche Beispiele sind Methylmethansulfonat (MMS) und Ethylnitrosoharnstoff (ENU).
Typische Beispiele für methylierte Basen sind 7-Methylguanin, 3-Methyladenin, 3-Methylguanin und 06-Methylguanin. Einige dieser Läsionen sind möglicherweise tödlich, da sie das Abwickeln von DNA während der Replikation und Transkription stören können. Die meisten sind auch indirekt mutagen; 0 6 -Methylguanin ist jedoch eine direkt mutagene Läsion, da es während der Replikation mit Thymin Basenpaare bilden kann.
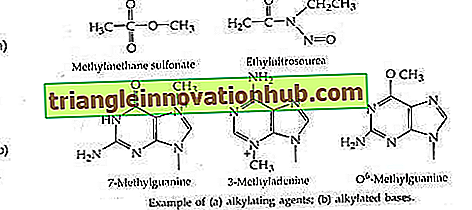
4. Sperrige Addukte:
Cyclobutanpyrimidindimere werden durch ultraviolettes Licht aus benachbarten Pyrimidinen an einem Strang durch Cyclisierung der doppelt gebundenen C5- und C6-Kohlenstoffatome jeder Base gebildet, um einen Cyclobutanring zu ergeben. Der resultierende Verlust der Basenpaarung mit dem Gegenstrang verursacht eine lokalisierte Denaturierung der DNA, die eine voluminöse Läsion erzeugt, die die Replikation und Transkription stören würde. Ein anderer Typ von Pyrimidindimer, das 6, 4-Photoprodukt, resultiert aus der Bildung einer Bindung zwischen C6 einer Pyrimidinbase und C4 der benachbarten Base.
Wenn das Kohlenteer-Karzinogen Benzo [a] pyren in der Leber durch Cytochrom P-450 metabolisiert wird, kann sich einer seiner Metaboliten (ein Diolepoxid) kovalent an die 2-Aminogruppe von Guaninresten anlagern. Viele andere aromatische Arylierungsmittel bilden mit DNA kovalente Addukte. Das Leberkarzinogen Aflatoxin B bindet auch kovalent an DNA.
DNA-Reparatur:
Die Reparatursysteme erkennen eine Vielzahl von Änderungen in der DNA, um eine Aktion einzuleiten. Eine Zelle kann mehrere Systeme haben, um mit DNA-Schäden fertig zu werden. Diese Systeme umfassen Folgendes: (1) Direktreparatur, (2) Exzisionsreparatur, (3) Fehlanpassungsreparatur, (4) tolerante Systeme und (5) Abrufsysteme.
1. Direkte Reparatur:
Die Umkehrung oder einfache Entfernung des Schadens an der DNA ist als direkte Reparatur bekannt, z. B. Entfernung der kovalenten Bindungen zwischen den zwei 4 und zwei 5 Kohlenstoffatomen der beiden Thyminreste, die an der Bildung von Thymin-Dimeren beteiligt sind.
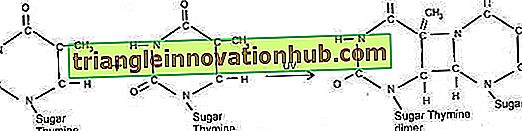
Thymin-Dimere werden im Allgemeinen durch UV-Bestrahlung gebildet und stören die Replikation und Transkription. Kelner (1949) beobachtete, dass eine große Anzahl von Bakterien große Dosen von UV-Strahlung überleben konnte; wenn sie einer intensiven sichtbaren Lichtquelle ausgesetzt waren.
Dieses Phänomen wird als Photoreaktivierung bezeichnet. Später wurde gezeigt, dass ein bestimmtes Enzym während der UV-Bestrahlung selektiv an die bakterielle DNA gebunden wird. Während des Photoreaktivierungsprozesses wird das Enzym durch sichtbares Licht aktiviert. Es spaltet die Thymin-Dimere und stellt sie so in ihre ursprüngliche Form zurück. Dieser Reparaturprozess ist enzymvermittelt und lichtabhängig.
2. Exzisionsreparatur:
Bei diesem Reparaturmechanismus wird das beschädigte oder veränderte Segment des DNA-Strangs herausgeschnitten und an seiner Stelle ein neues DNA-Stück synthetisiert. Obwohl Exzisionsreparatursysteme in ihrer Spezifität variieren, umfasst der Hauptpfad die folgenden drei Schritte:
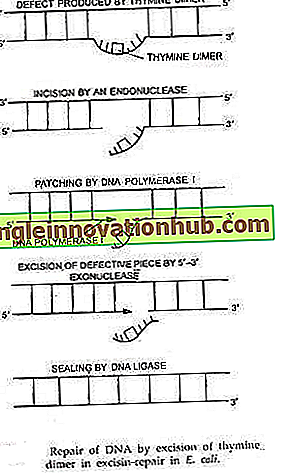
(i) Anerkennung und Schnitt:
Der beschädigte / veränderte Abschnitt eines DNA-Strangs wird von einer Endonuklease erkannt; Dieses Enzym schneidet dann den betroffenen Strang auf beiden Seiten des Schadens.
(ii) Exzision:
Eine 5 '-> 3'-Exonuklease (DNA-Polymerase I) verdaut den beschädigten / veränderten Abschnitt; Dies erzeugt eine einzelsträngige Region in der DNA-Doppelhelix.
(iii) Synthese:
In diesem Schritt dient der durch Exzision erzeugte einzelsträngige Bereich als Matrize für eine DNA-Polymerase, die den Ersatz für das ausgeschnittene Segment synthetisiert. DNA-Ligase besiegelt dann den Rest, der nach der Synthese des Ersatzes für den herausgeschnittenen Abschnitt übrig bleibt.
In E. coli ist die Exzision höchstwahrscheinlich auf die 3 '-> 5'-Exonuklease-Aktivität der DNA-Polymerase I zurückzuführen, während die Synthese durch die 5' -> 3'-Polymerase-Aktivität desselben Enzyms erfolgt. Die Exzisionsreparatursysteme sind sowohl bei Prokaryoten als auch bei Eukaryoten recht häufig. Einige dieser Systeme erkennen eine allgemeine Schädigung der DNA, während andere sehr spezifisch sind, z. B. entfernen AP-Endonukleasen die Ribosereste von den Depurinationsstellen. Die Exzisionsreparatur umfasst unterschiedliche DNA-Längen und ist in die folgenden drei Klassen unterteilt:
(a) Sehr kurze Patch-Reparatur (VSP):
Dieses System befasst sich mit der Reparatur von Unstimmigkeiten zwischen bestimmten Basen.
(b) Kurze Patch-Reparatur:
In diesem System wird ein etwa 20 Basen langer DNA-Strang herausgeschnitten, und die Schäden werden durch uvr-Gene (uvr, A, B, C von E. coli) repariert, die für Komponenten einer Reparaturendonuklease kodieren. Ein weiteres Enzym, uvr D, wird auch für die Helicase-Aktivität benötigt.
(c) Lange Patch-Reparatur:
Dieses System beinhaltet die Entfernung von ungefähr 1500 Basen langen Segmenten, aber manchmal kann das ausgeschnittene Segment> 9000 Basen sein. Es ist viel seltener und muss durch Schäden induziert werden, die die Replikation blockieren. In E. coli umfasst dieses System auch die uvr-Gene und die DNA-Polymerase I.
3. Nichtübereinstimmung reparieren:
Dies beinhaltet die Korrektur von Fehlpaarungen oder die Paarung zwischen Basen, die nicht komplementär sind. Fehlpaarungen können entweder (a) während der Replikation oder (b) aufgrund von Basenänderungen (z. B. Desaminierung von Cytosin zu Uracil) auftreten und zu strukturellen Verzerrungen in der Doppelhelix der DNA führen. Diese Änderungen werden in E. coli durch das nachstehend beschriebene, sehr kurze Patch-Exzisionsreparatursystem behandelt.
Mismatch Repair System in E. coli:
Wenn ein Basenpaar wie in GC -> GT nicht übereinstimmt, kann es theoretisch repariert werden, um entweder Wildtyp (GC) oder einen Mutantentyp (AT) zu erzeugen. Daher muss das Reparatursystem zwischen alten und neuen Strängen unterscheiden und nur den neuen Strang reparieren, um den Wildtyp wiederherzustellen.
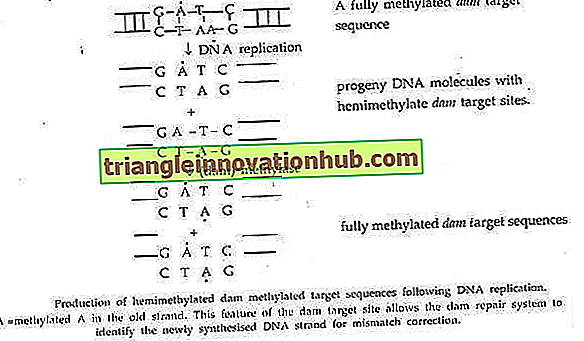
Dies geschieht durch ein sehr kurzes Patch-Exzisionsreparatursystem und erfordert vier Proteine, nämlich Mut L, Mut S, Mut U und Mut H, die in E. coli durch die Gene Mut L, Mut S, Mut U und Mut H codiert werden. Die Fehlpaarungsfehler Die während der Replikation erzeugten Produkte werden durch das Dammreparatursystem korrigiert.
Das Gen dam von E. coli produziert eine Methylase, die Adenin der Sequenz GATC an beiden DNA-Strängen methyliert. Die Replikation einer vollständig methylierten GATC-Sequenz ergibt eine hemimethylierte Sequenz, in der die A-Reste in den neu synthetisierten Strängen nicht methyliert sind.
Der nicht methylierte Zustand dieser Zielsequenz wird verwendet, um den neuen Strang zu identifizieren; Die Basen um den Ort der Fehlpaarung im neuen Strang werden herausgeschnitten und ein Ersatz hergestellt. Das System umfasst die Produkte der Gene mut L, mut S, mut H und uvr D.
Retrieval Systems Reparaturen:
Diese Systeme werden auch als "Reparatur nach der Replikation" oder "Rekombination" bezeichnet. In E. coli basiert dieses Reparatursystem auf dem rec A-Gen, das das Rec A-Protein produziert. Rec Ein Protein fungiert beim Austausch von Strängen zwischen DNA-Molekülen während der genetischen Rekombination und auch beim Einzelstrangaustausch während der Rekombinationsreparatur. Es scheint zwei rec A-Wege zu geben, einen mit den rec B, C-Genen und den anderen mit rec F.
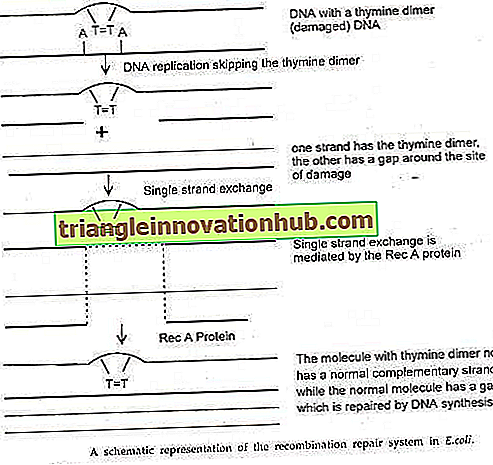
Dieses Reparatursystem funktioniert, wenn eine strukturelle Verzerrung die Replikation an der beschädigten Stelle blockiert. Zum Beispiel können E. coli-Mutanten, denen die Exzisionsreparatur fehlt, keine Thymin-Dimere entfernen. In einer solchen Situation wird die Replikation normalerweise bis zu den beschädigten Standorten durchgeführt. Die DNA-Polymerase stoppt die Replikation des betroffenen Strangs und initiiert die Replikation erneut, wobei sie an der beschädigten Stelle vorbeigeht.
Der komplementäre Strang wird normalerweise an der beschädigten Stelle im anderen Strang repliziert. Daher ergibt die Replikation eine normale Nachkommenschaft eines DNA-Moleküls und ein Molekül mit dem Thymin-Dimer in einem Strang und einer langen Lücke in seinem komplementären Strang. Das DNA-Molekül der Nachkommenschaft mit Thymin-Dimer würde verloren gehen, wenn es nicht durch Füllen der Lücke in einem seiner Stränge repariert wird.
Dies wird durch Einzelstrangaustausch zwischen den beiden Nachkommen-DNA-Molekülen erreicht; Die Austauschregion füllt die Lücke und wird durch das Rec A-Protein induziert. Infolge dieses Austausches weist das normale Nachkommenmolekül eine einzelsträngige Region auf, die eine Lücke aufweist. Diese Lücke wird durch DNA-Synthese behoben.
Toleranzsystem:
Diese Systeme befassen sich mit den Schäden, die die normale Replikation an der beschädigten Site blockieren, indem sie die Replikation der beschädigten Sites mit einer hohen Fehlerhäufigkeit ermöglichen. Diese Systeme können besonders wichtig bei Eukaryoten sein, bei denen die Genomgröße sehr groß ist und eine vollständige Behebung des Schadens eher unwahrscheinlich ist.